
(Unig – Itaperuna – 2018-2)
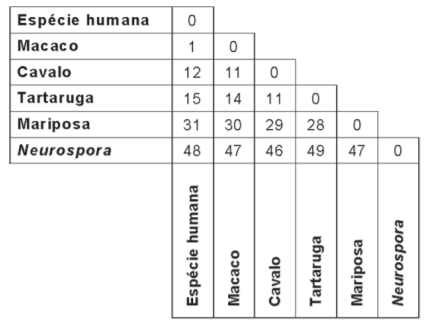
A tabela apresenta dados relativos ao número de diferenças na sequência de aminoácidos da cadeia polipeptídica do citocromo c em algumas espécies biológicas.
A partir da análise dos dados sob uma perspectiva da evolução molecular, conclui-se:
01) O grau de parentesco entre duas espécies é diretamente proporcional ao número de aminoácidos diferentes nos
citocromos.
02) As diferenças identificadas podem ser explicadas como decorrência de mutações em que houve perda de nucleotídeos no gene original.
03) A porcentagem em que ocorrem os diferentes aminoácidos na construção da proteína é a mesma em todos os citocromos analisados.
04) As variações na estrutura primária, entre as moléculas de citocromo c expressam as diferenças gênicas entre as espécies analisadas.
05) As diferenças entre o citocromo c do chimpanzé e o do homem resultam na alteração de três códons no RNAm do homem em relação ao citocromo do chimpanzé.
RESOLUÇÃO:
Entre o homem e o macaco há apenas 1 aminoácido diferente na molécula de citocromo C, o que indica a ocorrência de apenas 1 códon (sequência de 3 bases consecutivas do RNA mensageiro) diferente no gene do homem e do macaco, envolvido com a síntese do citocromo. A sequência linear de aminoácidos de uma molécula proteica é a estrutura primária da proteína, que é geneticamente determinada. Dessa forma, diferenças na estrutura primária refletem diferenças no gene que codifica a proteína em questão.
Quanto menor a diferença bioquímica entre duas biomoléculas, maior o grau de parentesco entre elas.
Resp.: 04




Seja o primeiro a comentar